精品推荐 | 微球菌核酸酶(MNase),告别超声损伤,精准实现染色质片段化,助力染色质免疫沉淀实验再升级!
微球菌核酸酶简介
微球菌核酸酶(Micrococcal Nuclease, MNase),也被称为Micrococcal Endonuclease或S7 Nuclease,是一种来源于金黄色葡萄球菌(Staphylococcus aureus)的DNA 和 RNA 核酸内切酶。MNase酶在pH7-10和Ca2+的条件下可降解单链、双链、线状、环状等多种形式的DNA或RNA的5'磷酸键,并产生3'磷酸末端的单核苷酸和寡核苷酸,常用于染色质免疫沉淀实验中的染色质片段化!
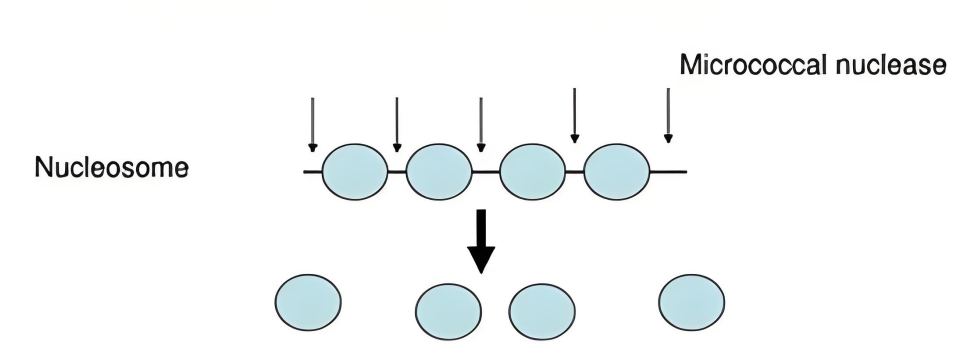
图1. Micrococcal Nuclease作用原理[1]
在染色质免疫沉淀实验中,MNase酶能够特异性消化核小体间连接区域的裸露DNA,而核小体核心颗粒中的DNA因受组蛋白的保护,可抵抗酶解作用,从而能完整保留与目标蛋白结合的DNA片段。相较于传统的超声波片段化方法(随机打断染色质,易损伤蛋白-DNA复合物),MNase酶切法具有高特异性与温和的酶切特性,能显著提升目标DNA的富集效率与实验分辨率(图2)。
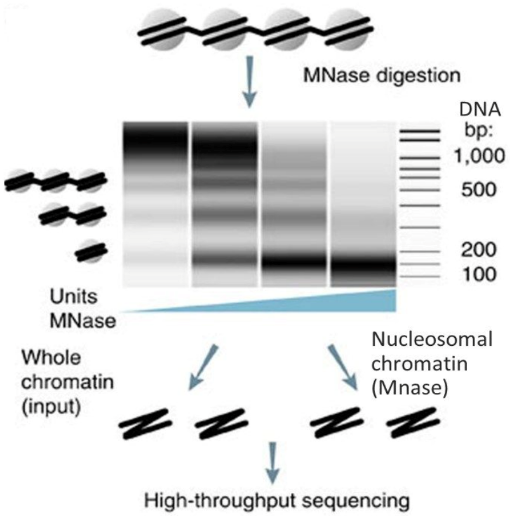
图2. MNase酶切割效果[2]
目前广泛应用的Ultra-Low-Input Micrococcal Nuclease-based Native ChIP (ULI-NChIP)[3] 是一种适用于极低细胞投入量的ChIP-seq新技术,该技术便是利用MNase酶解替代传统的超声法进行染色质的片段化。ULI-NchIP适用于微量实验材料, 可以从103 个细胞起始构建高质量测序文库, 适用于组蛋白修饰情况研究。
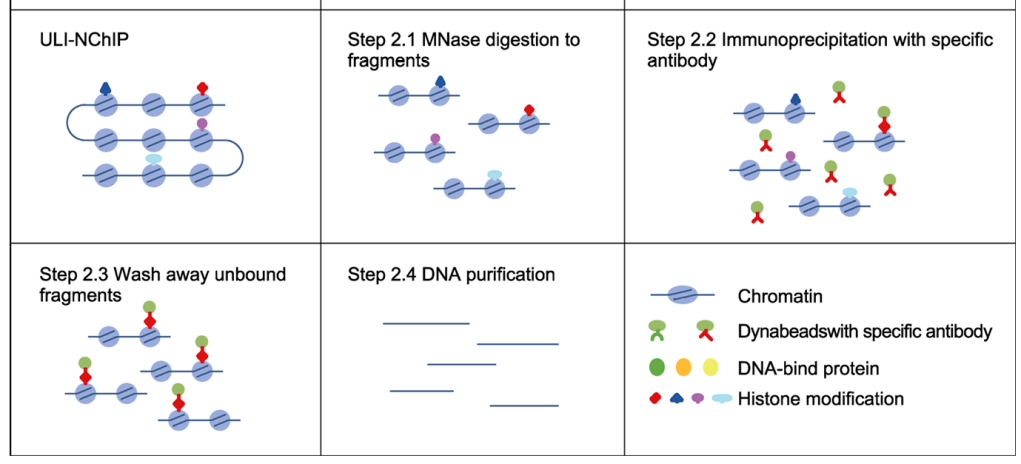
图3. ULI-NchIP-seq实验流程图[4]
性能展示
Micrococcal Nuclease (Cat#14547ES)
DNA底物切除能力与进口品牌A*一致
分别使用翌圣的Micrococcal Nuclease与进口品牌A*的同类产品切除1μg λ DNA底物,50 μL体系,37℃反应15min。琼脂糖凝胶电泳检测结果表明,翌圣Micrococcal Nuclease与进口品牌A*的切除效果一致。
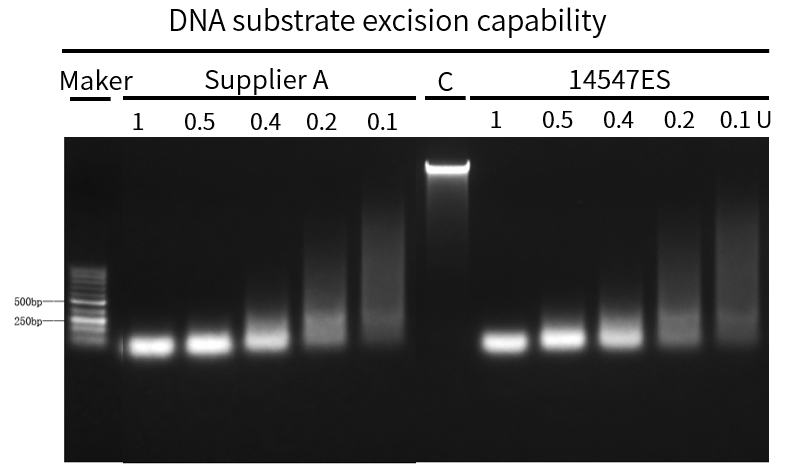
图4. DNA底物切除能力
无蛋白酶残留检测
将不同批次的10000 U Micrococcal Nuclease分别与相应的底物蛋白在37℃孵育16 h,琼脂糖凝胶电泳检测结果表明,翌圣Micrococcal Nuclease无蛋白酶残留。这一特性有效避免了因蛋白酶残留导致的蛋白质非特异性降解,确保实验中核酸与蛋白质互作研究结果的准确性。
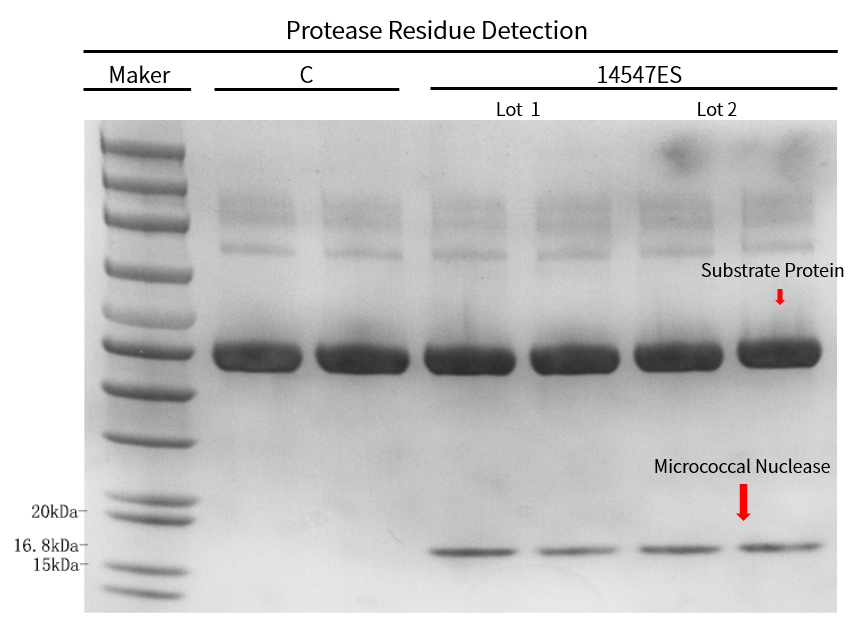
图5. 蛋白酶检测
E. coli基因组DNA残留< 1 copy/2000 U
对不同批次的Micrococcal Nuclease进行E. coli gDNA残留检测,结果显示翌圣Micrococcal Nuclease宿主基因组DNA残留低于1 copy/2000 U。
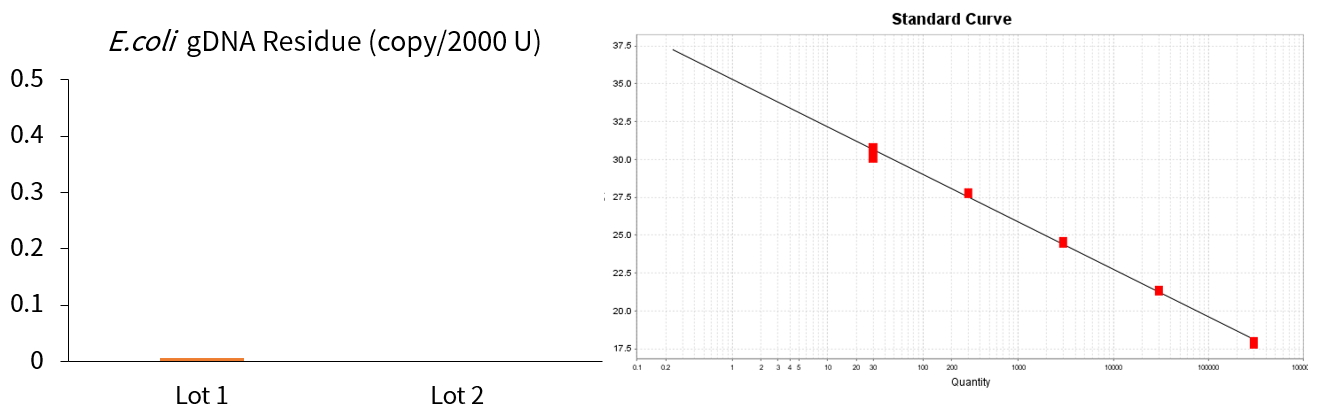
图6. E. coli g DNA残留检测
产品推荐
|
产品名称 |
产品货号 |
规格 |
|
14547ES |
320/1600 KU |
|
|
12927ES |
8/24/96 T |
|
|
12601ES |
1/5/60/450 mL |
|
|
12642ES |
100/500 T |
参考文献
[1] Alexander M, Heppel LA, Hurwitz J. The purification and properties of micrococcal nuclease. Journal of Biological Chemistry. 1961;236:3014-3019
[2] Furey,Terrence S . ChIP-seq and beyond: new and improved methodologies to detect and characterize protein-DNA interactions.[J]. Nature Reviews Genetics, 2012, 13(12):840-52.
[3] Brind'Amour J, Liu S, Hudson M, Chen C, Karimi MM, Lorincz MC. An ultra-low-input native ChIP-seq protocol for genome-wide profiling of rare cell populations. Nat Commun. 2015 Jan 21;6:6033. doi: 10.1038/ncomms7033. PMID: 25607992.
[4] 王泓力,焦雨铃.染色质免疫共沉淀实验方法[J].植物学报, 2020, 55(4):6.DOI:10.11983/CBB20076.
